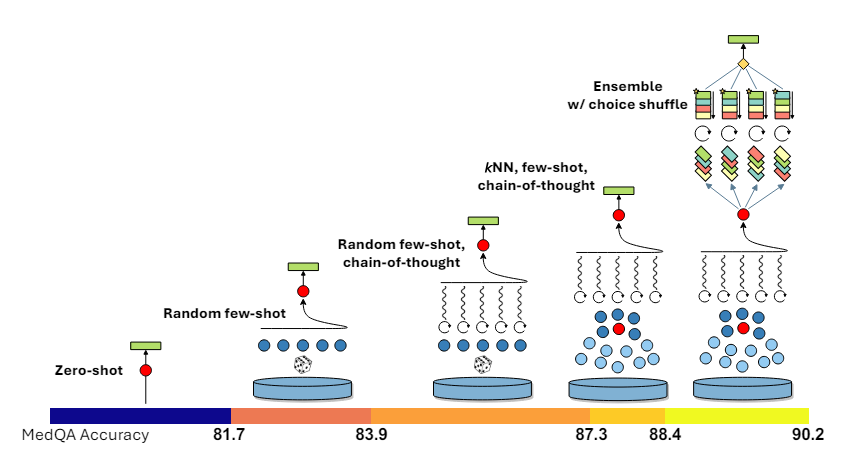
ahora estamos corriendo elección de montaje de mezcla Al mezclar el orden de las opciones de respuesta para cada pregunta del examen, creamos múltiples variaciones de la misma pregunta. Luego se le pide al LLM que utilice estas variantes, junto con los ejemplos correspondientes, para generar pasos de razonamiento y una respuesta para cada variante. Finalmente, realizamos una votación mayoritaria sobre las predicciones de todas las variantes y seleccionamos la predicción final.
El código para esta implementación se puede encontrar aquí. enlace al repositorio de github.
Usamos MedQA [6] Conjunto de datos para la implementación y evaluación de Medprompt. Primero definimos funciones auxiliares para analizar archivos jsonl.
def write_jsonl_file(file_path, dict_list):
"""
Write a list of dictionaries to a JSON Lines file.Args:
- file_path (str): The path to the file where the data will be written.
- dict_list (list): A list of dictionaries to write to the file.
"""
with open(file_path, 'w') as file:
for dictionary in dict_list:
json_line = json.dumps(dictionary)
file.write(json_line + '\n')
def read_jsonl_file(file_path):
"""
Parses a JSONL (JSON Lines) file and returns a list of dictionaries.
Args:
file_path (str): The path to the JSONL file to be read.
Returns:
list of dict: A list where each element is a dictionary representing
a JSON object from the file.
"""
jsonl_lines = []
with open(file_path, 'r', encoding="utf-8") as file:
for line in file:
json_object = json.loads(line)
jsonl_lines.append(json_object)
return jsonl_lines
Implementación de CoT autogenerado
Para nuestra implementación, utilizamos el conjunto de formación MedQA. Implementamos un mensaje CoT de disparo cero y abordamos todas las preguntas de capacitación. Utilizamos GPT-4o En nuestra implementación generamos para cada pregunta el CoT y la respuesta correspondiente. Definimos un mensaje basado en la plantilla proporcionada en el documento Medprompt.
system_prompt = """You are an expert medical professional. You are provided with a medical question with multiple answer choices.
Your goal is to think through the question carefully and explain your reasoning step by step before selecting the final answer.
Respond only with the reasoning steps and answer as specified below.
Below is the format for each question and answer:Input:
## Question: {{question}}
{{answer_choices}}
Output:
## Answer
(model generated chain of thought explanation)
Therefore, the answer is [final model answer (e.g. A,B,C,D)]"""
def build_few_shot_prompt(system_prompt, question, examples, include_cot=True):
"""
Builds the zero-shot prompt.Args:
system_prompt (str): Task Instruction for the LLM
content (dict): The content for which to create a query, formatted as
required by `create_query`.
Returns:
list of dict: A list of messages, including a system message defining
the task and a user message with the input question.
"""
messages = [{"role": "system", "content": system_prompt}]
for elem in examples:
messages.append({"role": "user", "content": create_query(elem)})
if include_cot:
messages.append({"role": "assistant", "content": format_answer(elem["cot"], elem["answer_idx"])})
else:
answer_string = f"""## Answer\nTherefore, the answer is {elem["answer_idx"]}"""
messages.append({"role": "assistant", "content": answer_string})
messages.append({"role": "user", "content": create_query(question)})
return messages
def get_response(messages, model_name, temperature = 0.0, max_tokens = 10):
"""
Obtains the responses/answers of the model through the chat-completions API.
Args:
messages (list of dict): The built messages provided to the API.
model_name (str): Name of the model to access through the API
temperature (float): A value between 0 and 1 that controls the randomness of the output.
A temperature value of 0 ideally makes the model pick the most likely token, making the outputs deterministic.
max_tokens (int): Maximum number of tokens that the model should generate
Returns:
str: The response message content from the model.
"""
response = client.chat.completions.create(
model=model_name,
messages=messages,
temperature=temperature,
max_tokens=max_tokens
)
return response.choices[0].message.content
También definimos funciones auxiliares para analizar el razonamiento y la opción de respuesta final de la respuesta LLM.
def matches_ans_option(s):
"""
Checks if the string starts with the specific pattern 'Therefore, the answer is [A-Z]'.Args:
s (str): The string to be checked.
Returns:
bool: True if the string matches the pattern, False otherwise.
"""
return bool(re.match(r'^Therefore, the answer is [A-Z]', s))
def extract_ans_option(s):
"""
Extracts the answer option (a single capital letter) from the start of the string.
Args:
s (str): The string containing the answer pattern.
Returns:
str or None: The captured answer option if the pattern is found, otherwise None.
"""
match = re.search(r'^Therefore, the answer is ([A-Z])', s)
if match:
return match.group(1) # Returns the captured alphabet
return None
def matches_answer_start(s):
"""
Checks if the string starts with the markdown header '## Answer'.
Args:
s (str): The string to be checked.
Returns:
bool: True if the string starts with '## Answer', False otherwise.
"""
return s.startswith("## Answer")
def validate_response(s):
"""
Validates a multi-line string response that it starts with '## Answer' and ends with the answer pattern.
Args:
s (str): The multi-line string response to be validated.
Returns:
bool: True if the response is valid, False otherwise.
"""
file_content = s.split("\n")
return matches_ans_option(file_content[-1]) and matches_answer_start(s)
def parse_answer(response):
"""
Parses a response that starts with '## Answer', extracting the reasoning and the answer choice.
Args:
response (str): The multi-line string response containing the answer and reasoning.
Returns:
tuple: A tuple containing the extracted CoT reasoning and the answer choice.
"""
split_response = response.split("\n")
assert split_response[0] == "## Answer"
cot_reasoning = "\n".join(split_response[1:-1]).strip()
ans_choice = extract_ans_option(split_response[-1])
return cot_reasoning, ans_choice
Ahora cubrimos las preguntas del conjunto de capacitación de MedQA. Obtenemos las respuestas de CoT y las respuestas a todas las preguntas y las almacenamos en una carpeta.
train_data = read_jsonl_file("data/phrases_no_exclude_train.jsonl")cot_responses = []
# os.mkdir("cot_responses")
existing_files = os.listdir("cot_responses/")
for idx, item in enumerate(tqdm(train_data)):
if str(idx) + ".txt" in existing_files:
continue
prompt = build_zero_shot_prompt(system_prompt, item)
try:
response = get_response(prompt, model_name="gpt-4o", max_tokens=500)
cot_responses.append(response)
with open(os.path.join("cot_responses", str(idx) + ".txt"), "w", encoding="utf-8") as f:
f.write(response)
except Exception as e :
print(str(e))
cot_responses.append("")
Ahora revisamos todas las respuestas generadas para verificar si son válidas y seguimos el formato de predicción definido en el mensaje. Eliminamos respuestas que no se ajusten al formato requerido. Luego comparamos las respuestas previstas con la verdad fundamental para cada pregunta y solo conservamos aquellas preguntas para las cuales las respuestas previstas coinciden con la verdad fundamental.
questions_dict = []
ctr = 0
for idx, question in enumerate(tqdm(train_data)):
file = open(os.path.join("cot_responses/", str(idx) + ".txt"), encoding="utf-8").read()
if not validate_response(file):
continuecot, pred_ans = parse_answer(file)
dict_elem = {}
dict_elem["idx"] = idx
dict_elem["question"] = question["question"]
dict_elem["answer"] = question["answer"]
dict_elem["options"] = question["options"]
dict_elem["cot"] = cot
dict_elem["pred_ans"] = pred_ans
questions_dict.append(dict_elem)
filtered_questions_dict = []
for item in tqdm(questions_dict):
pred_ans = item["options"][item["pred_ans"]]
if pred_ans == item["answer"]:
filtered_questions_dict.append(item)
Implementación del modelo KNN.
Después de procesar el conjunto de capacitación y obtener la respuesta de CoT para todas estas preguntas, ahora integramos todas las preguntas usando integración-de-texto-ada-002 de OpenAI.
def get_embedding(text, model="text-embedding-ada-002"):
return client.embeddings.create(input = [text], model=model).data[0].embeddingfor item in tqdm(filtered_questions_dict):
item["embedding"] = get_embedding(item["question"])
inv_options_map = {v:k for k,v in item["options"].items()}
item["answer_idx"] = inv_options_map[item["answer"]]
Ahora entrenamos un modelo KNN utilizando estas incorporaciones de preguntas. Esto actúa como un recuperador en el momento de la inferencia porque nos ayuda a recuperar puntos de datos similares del conjunto de entrenamiento que son más similares a la pregunta del conjunto de prueba.
import numpy as np
from sklearn.neighbors import NearestNeighborsembeddings = np.array([d["embedding"] for d in filtered_questions_dict])
indices = list(range(len(filtered_questions_dict)))
knn = NearestNeighbors(n_neighbors=5, algorithm='auto', metric='cosine').fit(embeddings)
Implementación de una lógica general para la mezcla dinámica de unos pocos toques y selecciones.
Ahora podemos ejecutar la inferencia. Tomamos una submuestra de 500 preguntas del conjunto de pruebas MedQA para nuestra evaluación. Para cada pregunta, recuperamos las 5 preguntas más similares del conjunto de capacitación utilizando el módulo KNN, junto con sus respectivos pasos de razonamiento CoT y respuestas previstas. Creamos un mensaje para algunas tomas usando estos ejemplos.
Para cada pregunta, también mezclamos el orden de las opciones 5 veces para crear diferentes variaciones. Luego utilizamos el mensaje construido en unos pocos movimientos para obtener la respuesta prevista para cada una de las variaciones con opciones mixtas.
def shuffle_option_labels(answer_options):
"""
Shuffles the options of the question.Parameters:
answer_options (dict): A dictionary with the options.
Returns:
dict: A new dictionary with the shuffled options.
"""
options = list(answer_options.values())
random.shuffle(options)
labels = [chr(i) for i in range(ord('A'), ord('A') + len(options))]
shuffled_options_dict = {label: option for label, option in zip(labels, options)}
return shuffled_options_dict
test_samples = read_jsonl_file("final_processed_test_set_responses_medprompt.jsonl")for question in tqdm(test_samples, colour ="green"):
question_variants = []
prompt_variants = []
cot_responses = []
question_embedding = get_embedding(question["question"])
distances, top_k_indices = knn.kneighbors([question_embedding], n_neighbors=5)
top_k_dicts = [filtered_questions_dict[i] for i in top_k_indices[0]]
question["outputs"] = []
for idx in range(5):
question_copy = question.copy()
shuffled_options = shuffle_option_labels(question["options"])
inv_map = {v:k for k,v in shuffled_options.items()}
question_copy["options"] = shuffled_options
question_copy["answer_idx"] = inv_map[question_copy["answer"]]
question_variants.append(question_copy)
prompt = build_few_shot_prompt(system_prompt, question_copy, top_k_dicts)
prompt_variants.append(prompt)
for prompt in tqdm(prompt_variants):
response = get_response(prompt, model_name="gpt-4o", max_tokens=500)
cot_responses.append(response)
for question_sample, answer in zip(question_variants, cot_responses):
if validate_response(answer):
cot, pred_ans = parse_answer(answer)
else:
cot = ""
pred_ans = ""
question["outputs"].append({"question": question_sample["question"], "options": question_sample["options"], "cot": cot, "pred_ans": question_sample["options"].get(pred_ans, "")})
Ahora evaluamos los resultados de Medprompt en el equipo de prueba. Para cada pregunta, tenemos cinco predicciones generadas por la lógica de conjunto. Tomamos la moda, o predicción más frecuente, para cada pregunta como predicción final y evaluamos el desempeño. Aquí son posibles dos casos límite:
- Se predicen dos opciones de respuesta diferentes dos veces cada una, sin un ganador claro.
- Hay un error con la respuesta generada, lo que significa que no tenemos una opción de respuesta prevista.
Para estos dos casos limitantes, consideramos que la pregunta está mal respondida por el LLM.
def find_mode_string_list(string_list):
"""
Finds the most frequently occurring strings.Parameters:
string_list (list of str): A list of strings.
Returns:
list of str or None: A list containing the most frequent string(s) from the input list.
Returns None if the input list is empty.
"""
if not string_list:
return None
string_counts = Counter(string_list)
max_freq = max(string_counts.values())
mode_strings = [string for string, count in string_counts.items() if count == max_freq]
return mode_strings
ctr = 0
for item in test_samples:
pred_ans = [x["pred_ans"] for x in item["outputs"]]
freq_ans = find_mode_string_list(pred_ans)
if len(freq_ans) > 1:
final_prediction = ""
else:
final_prediction = freq_ans[0]
if final_prediction == item["answer"]:
ctr +=1
print(ctr / len(test_samples))
Evaluamos el desempeño de Medprompt con GPT-4o en términos de precisión en el subconjunto de pruebas MedQA. Además, comparamos el rendimiento del aviso de disparo cero, el aviso de pocos disparos aleatorios y el aviso de pocos disparos aleatorios con CoT.
Observamos que los incentivos CoT Medprompt y Random Few-Shot funcionan mejor que los incentivos Zero y Few-Shot. Sin embargo, sorprendentemente, notamos que Random Few-Shot CoT supera nuestro rendimiento de Medprompt. Esto podría deberse a varias razones:
- El artículo original de Medprompt evaluó el desempeño de GPT-4. Observamos que GPT-4o supera significativamente a GPT-4T y GPT-4 en varias pruebas de texto (https://openai.com/index/bonjour-gpt-4o/), lo que indica que Medprompt podría tener un efecto menor en un modelo más potente como GPT-4o.
- Limitamos nuestra evaluación a 500 preguntas submuestreadas de MedQA. El artículo de Medprompt evalúa otros conjuntos de datos de Medical MCQA y la versión completa de MedQA. La evaluación de GPT-4o en las versiones completas de los conjuntos de datos podría brindar una mejor imagen del rendimiento general.
Medprompt es un marco interesante para crear procesos de solicitud sofisticados, particularmente para adaptar un LLM general a un dominio específico sin necesidad de realizar ajustes. También destaca las consideraciones involucradas al elegir entre solicitud y ajuste para diversos casos de uso. Es importante explorar hasta qué punto se puede impulsar la licitación para mejorar el desempeño del LLM, ya que proporciona una alternativa rentable y rentable al ajuste.
[1] Nori, H., Lee, YT, Zhang, S., Carignan, D., Edgar, R., Fusi, N.,… y Horvitz, E. (2023). ¿Pueden los modelos básicos de propósito general superar a los modelos de ajuste de propósito específico? Estudio de caso en medicina. preimpresión de arXiv arXiv:2311.16452. (https://arxiv.org/abs/2311.16452)
[2] Wei, J., Wang, X., Schuurmans, D., Bosma, M., Xia, F., Chi, E.,… y Zhou, D. (2022). La cadena de pensamiento provoca el razonamiento en los principales patrones lingüísticos. Avances en los sistemas de procesamiento de información neuronal., 3524824–24837. (https://openreview.net/pdf?id=_VjQlMeSB_J)
[3] Gekhman, Z., Yona, G., Aharoni, R., Eyal, M., Feder, A., Reichart, R. y Herzig, J. (2024). ¿La optimización de los LLM sobre nuevos conocimientos promueve las alucinaciones? preimpresión de arXiv arXiv:2405.05904. (https://arxiv.org/abs/2405.05904)
[4] Singhal, K., Azizi, S., Tu, T., Mahdavi, SS, Wei, J., Chung, HW,… y Natarajan, V. (2023). Los grandes modelos de lenguaje codifican el conocimiento clínico. Naturaleza, 620(7972), 172–180. (https://www.nature.com/articles/s41586-023-06291-2)
[5] Singhal, K., Tu, T., Gottweis, J., Sayres, R., Wulczyn, E., Hou, L.,… y Natarajan, V. (2023). Hacia respuestas a preguntas médicas de nivel experto con grandes modelos de lenguaje. preimpresión de arXiv arXiv:2305.09617. (https://arxiv.org/abs/2305.09617)
[6] Jin, D., Pan, E., Oufattole, N., Weng, WH, Fang, H. y Szolovits, P. (2021). ¿Qué enfermedad padece este paciente? un conjunto de datos de respuestas a preguntas abiertas a gran escala de exámenes médicos. Ciencia aplicada, 11(14), 6421. (https://arxiv.org/abs/2009.13081) (El conjunto de datos original se publica bajo una licencia del MIT)