Así como ChatGPT genera texto prediciendo la palabra más probable a seguir en una secuencia, un nuevo inteligencia artificial El modelo (IA) puede escribir desde cero nuevas proteínas que no existen de forma natural.
Los científicos utilizaron el nuevo modelo, ESM3, para crear una nueva proteína fluorescente que comparte sólo el 58% de su secuencia con proteínas fluorescentes naturales, dijeron en un estudio preimpreso publicado el 2 de julio. base de datos bioRxivRepresentantes de EvolutionaryScale, una empresa fundada por ex investigadores de Meta, también presentaron los detalles el 25 de junio en un declaración.
El equipo de investigación publicó un versión pequeña del modelo bajo una licencia no comercial y pondrá la versión ampliada del modelo a disposición de investigadores comerciales. Según EvolutionaryScale, la tecnología podría resultar útil en áreas que van desde el descubrimiento de fármacos hasta el diseño de nuevos productos químicos para la degradación del plástico.
ESM3 es un modelo de lenguaje grande (LLM) similar al GPT-4 de OpenAI, que impulsa el chatbot ChatGPT, y los científicos entrenaron su versión más grande con 2,78 mil millones de proteínas. Para cada proteína, extrajeron información sobre la secuencia (el orden de los bloques de aminoácidos que forman la proteína), la estructura (la forma tridimensional plegada de la proteína) y la función (lo que hace la proteína). Enmascararon aleatoriamente fragmentos de información sobre estas proteínas y le pidieron a ESM3 que predijera los fragmentos faltantes.
Desarrollaron este modelo a partir de una investigación que el mismo equipo estaba realizando cuando aún estaba en Meta. En 2022, ellos anunció EMSFold — un precursor de ESM3 que predijo estructuras de proteínas microbianas desconocidas. Ese año, Alfabeto mente profunda también estructuras proteicas predichas por 200 millones de proteínas.
Relacionado: El programa de IA AlphaFold3 de DeepMind puede predecir la estructura de cada proteína del universo y mostrar cómo funcionan
Los científicos luego señalaron que hay Los límites de las predicciones de estos modelos de IA. y que las predicciones de proteínas deben verificarse. Pero estos métodos aún pueden acelerar significativamente la búsqueda de estructuras proteicas, porque la alternativa es utilizar rayos X para mapear las estructuras proteicas una por una, lo cual es lento y costoso.
ESM3, sin embargo, va más allá de simplemente predecir proteínas existentes. Utilizando información recopilada de 771 mil millones de piezas únicas de estructura, función y secuencia, el modelo puede generar nuevas proteínas con funciones particulares. Ha sido descrito como un “momento ChatGPT para la biología” por uno de los contribuyentes a EvolutionaryScale.
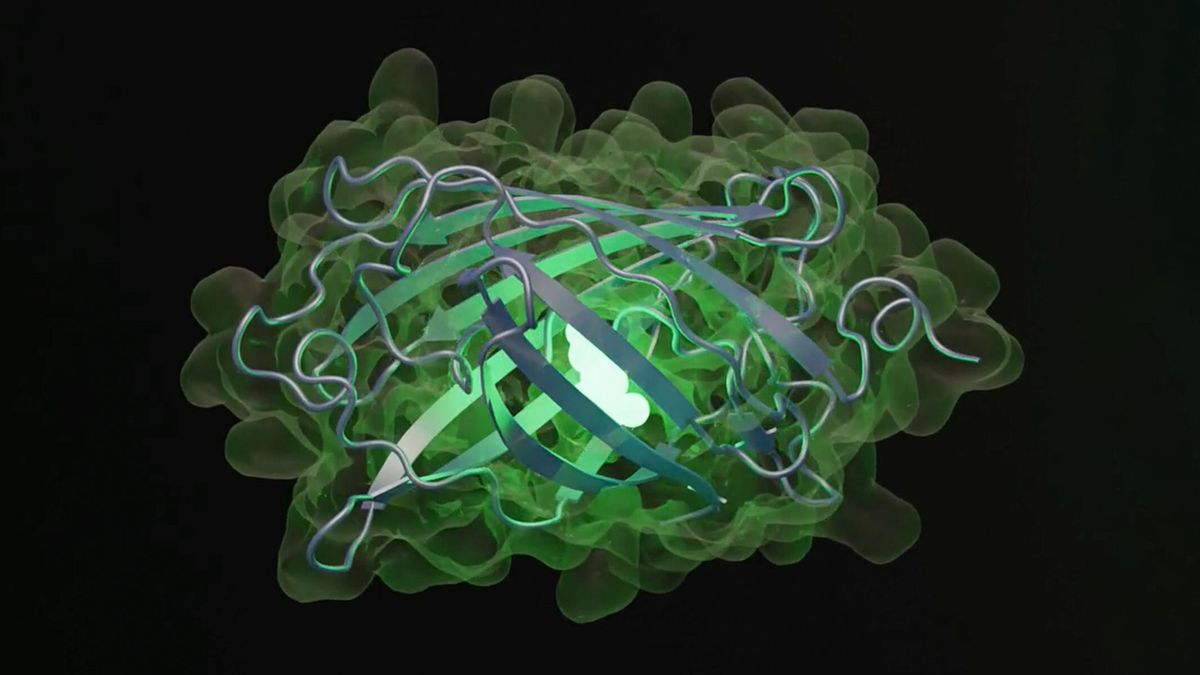
En el nuevo estudio, los investigadores interrogaron al modelo para generar una nueva proteína fluorescente, un tipo de proteína que captura la luz y la emite en una longitud de onda más larga, haciéndola brillar con un nuevo tono de verde. Estas proteínas son importantes para los investigadores biológicos que las asocian con las moléculas que desean estudiar para rastrearlas y obtener imágenes; su descubrimiento y desarrollo ganaron un premio Premio Nobel de Química En 2008.
El modelo generó 96 proteínas con secuencias y estructuras que probablemente produzcan fluorescencia. Luego, los investigadores eligieron el que tenía menos secuencias en común con las proteínas naturalmente fluorescentes. Aunque esta proteína era 50 veces menos brillante que las proteínas fluorescentes verdes naturales, ESM3 generó otra iteración que condujo a nuevas secuencias que aumentaron el brillo, y el resultado fue una proteína fluorescente verde diferente a cualquier otra encontrada en la naturaleza, apodada «esmGPF». Estas iteraciones, realizadas en momentos por IA, requerirían 500 millones de años de evolución para completarse, estimó el equipo de EvolutionaryScale.

“En la actualidad, todavía no entendemos cómo se comportan las proteínas, especialmente aquellas que son ‘nuevas para la ciencia’, cuando se introducen en un sistema vivo, pero este es un nuevo paso interesante que nos permite abordar la biología sintética de una manera nueva. Los modelos de IA como ESM3 permitirán el descubrimiento de nuevas proteínas que las limitaciones de la selección natural nunca permitirían, creando innovaciones en la ingeniería de proteínas que la evolución no puede permitir. Es apasionante. Sin embargo, la afirmación de que es posible simular 500 millones de años de evolución se centra únicamente en proteínas individuales, lo que ignora los numerosos pasos de la selección natural que crean la diversidad de vida que conocemos hoy. La ingeniería de proteínas impulsada por la IA es intrigante, pero no puedo evitar pensar que podemos estar demasiado confiados al suponer que podemos burlar los complejos procesos perfeccionados por millones de años de selección natural. »